一、任务
通过分子动力学模拟来探索不同理化性质 MPs(聚酯 PET、聚乙烯 PE、聚 丙烯酸酯 PP)与细胞膜的直接相互作用。
经文献调研,PET 微塑料包含多条聚对苯二甲酸乙二醇酯链,化学式为(C10H8O4)n/COC6H4COOCH2CH2O,每条链含约 100 个对苯二甲酸单体单元和 约 100 个乙二醇单体单元;PE 微塑料包含多条聚乙烯链,化学式为 (C2H4)n, 每条链含约 100 个乙烯单体单元;PP微塑料包含多条聚丙烯链,化学式为 (C3H6)n,每条链含约 100 个丙烯单体单元。
也就是
①PET100(C996H792O400)(邻苯二甲酸)
②PE100(C202H404) ethylene(乙烯)
③PP100(C301H602) propylene(丙烯)
通过 CHARMM-GUI 网站上的膜构建器获得 DPPC 双分子层。所得的脂 质双层由 256 个 DPPC 分子组成,膜的上下层对称分布。DPPC 双层的上小叶 和下小叶分别具有 128 个 DPPC 分子。DPPC 分子的亲水性头部向外,疏水性 尾部向内。DPPC 双分子层的初始结构在 GROMACS 53A6 力场下通过 100 ns 的分子动力学模拟进行结构优化。
一.数据生成charmm-gui/Materials Studio
https://charmm-gui.org/?doc=input/polymer
这里分为两种获取方式,因为gui网站生成蛋白质类物质特别的方便,因此DPCC的生成我们选择gui网站下载。聚合物方面我们选择选用MS进行生成并优化,因为gui网站下载的聚合物没有进行优化,在后续拓扑中缺少二面角等信息,因此我们聚合物选择MS进行生成。
1. DPCC生成
参考:https://www.bilibili.com/read/cv15787466/
到这个网站上下载数据
在这里我们要生成256个DPPC分子组成的的模拟双分子层,膜的上下层对称分布。
点击这里(这个注册需要一点时间,需要你填好信息以后,官方对你的信息进行审核,审核过程1天到半个月不等,官方审核完会把你的账号密码发送到你的邮箱里)
a 使用说明
依次点击Input Generator –> Membrane Builder –> Bilayer Builder,就会出现下图,网页下拉就能看到Protein/Membrane System以及Membrane Only System两个模块。
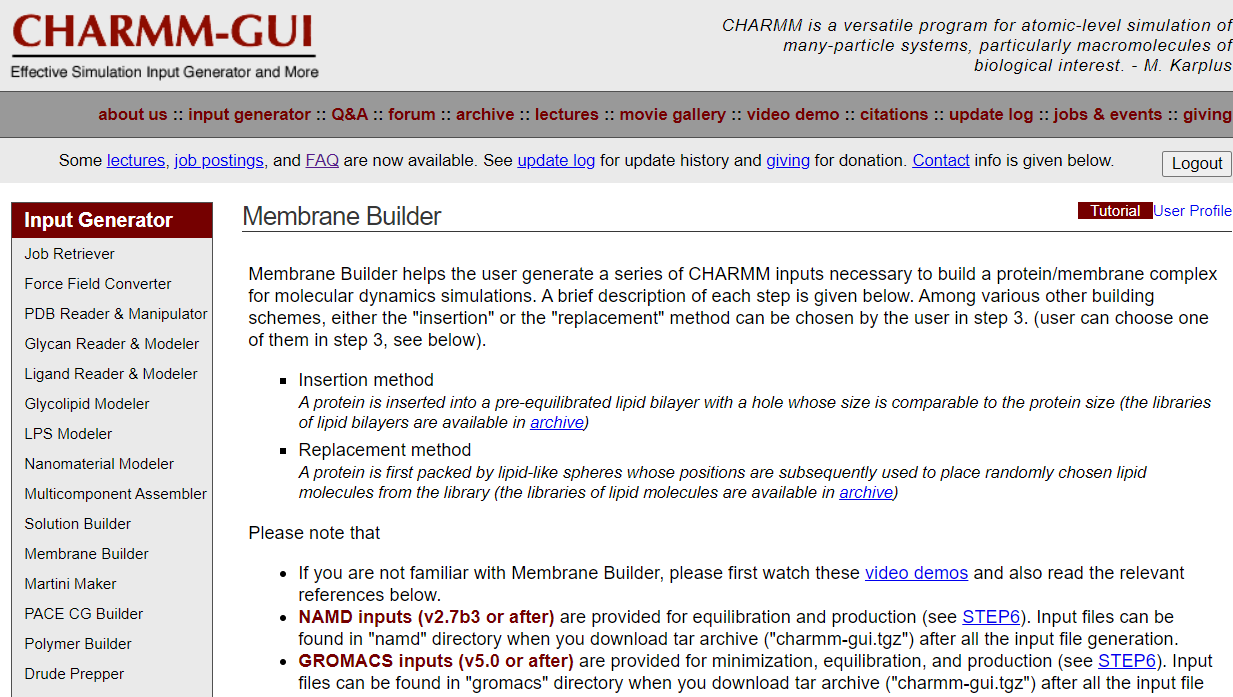
Membrane Builder可以帮助用户生成一系列charmm输入,以构建用于分子动力学模拟的蛋白质/膜复合物。
- 注意在Please note that下面有三句话:
1.a homogeneous lipid bilayer can be built with DMPC, DPPC, DOPC, POPC, DLPE, and POPE.(DMPC、DPPC、DOPC、POPC、DLPE和POPE可形成均质脂质双分子层)
2.a heterogeneous lipid bilayer can be built with 434 different lipid molecules (see lipid list).(非均相脂质双分子层可以由434种不同的脂质分子组成(看lipid list))
3.the heterogenous Membrane Builder can be used for a homogeneous lipid bilayer (only using the replacement method).(非均质膜构建模块可用于均质脂质双分子层构建)
b 构建DPPC膜
1)点击Membrane Only System
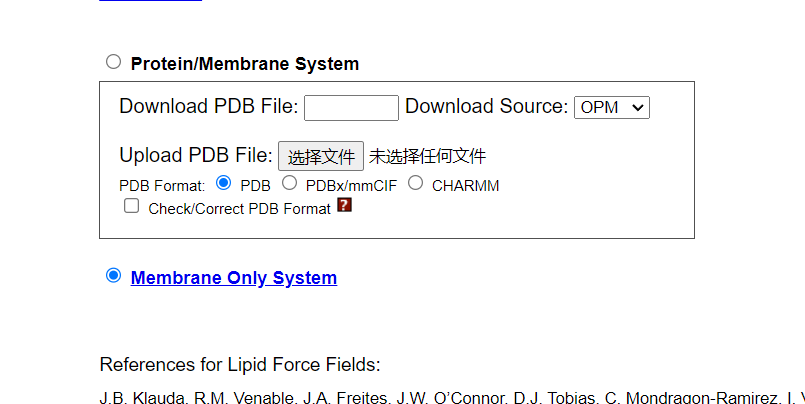
2)点击Next Step: Select Lipids,就会出现下图所示页面:
出现一行红字:”Homogeneous Lipid”选项不在支持,可以使用”Heterogeneous Lipid”来构建Homogeneous Lipid。所以我们直接
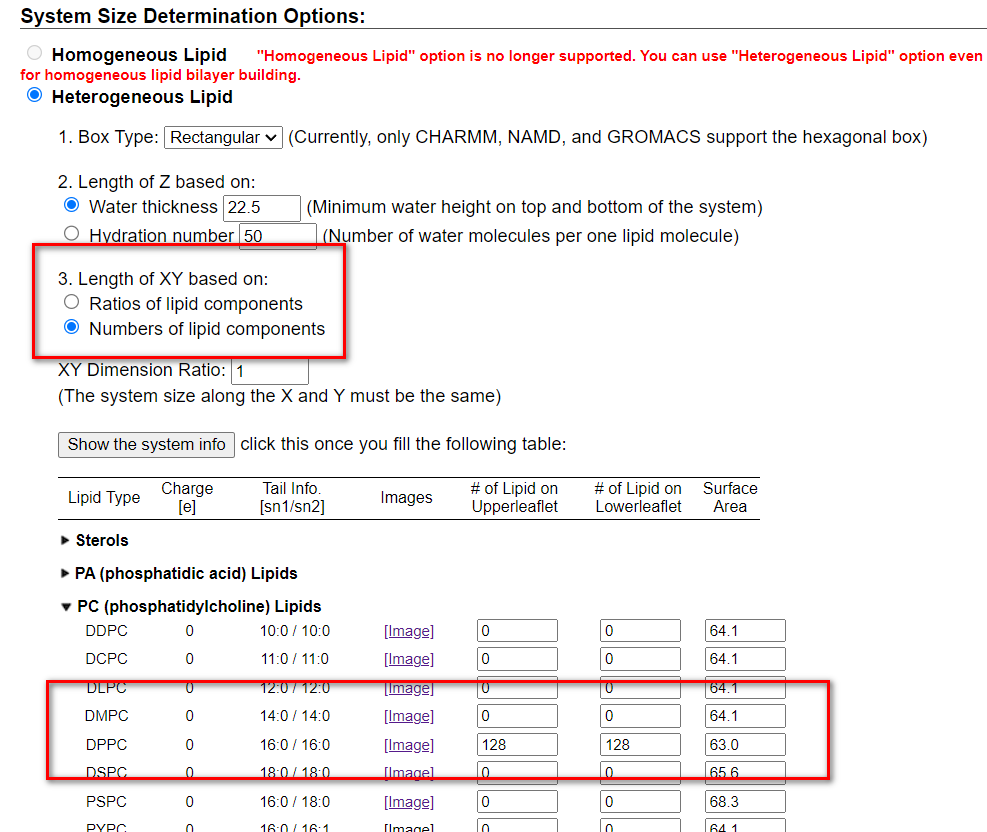
3)点击Heterogeneous Lipid,出现下图
一般不需要变动,因为我们需要构建256个DPPC分子,因此我们在3.中选择Number of lipid components ,上面指的是比例,如果选择比例,我们在下面选择构成比例时就要保证加起来等于100。我们这里选择DPPC分子,上下各128个分子。然后点击show the system info然后一直下一步就行
2. MS生成优化后的聚合物
使用 Materials Studio8.0 软件生 成 并 分 别 构 建 优化 PET100(C996H792O400)、PE100(C202H404)和 PP100(C301H602)的结构。
这里只展示PE的生成过程
a. 开始构建
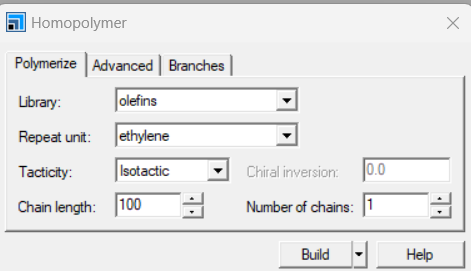
聚合度为100的PE链
%20d0e4e4faba0447f6895cb79f67055395/Untitled%205.png)
b对单链进行结构优化
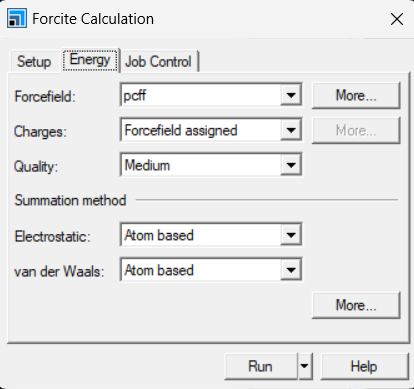
选择Forcite模块中的calculation。Setup栏选择Geometry Optimization, 选择Algorithm类型,针对聚合物单链,本人一般简单的smart,Energy选择力场,针对PE我选用pcff力场,Job control选择并行数,一般调到最大。
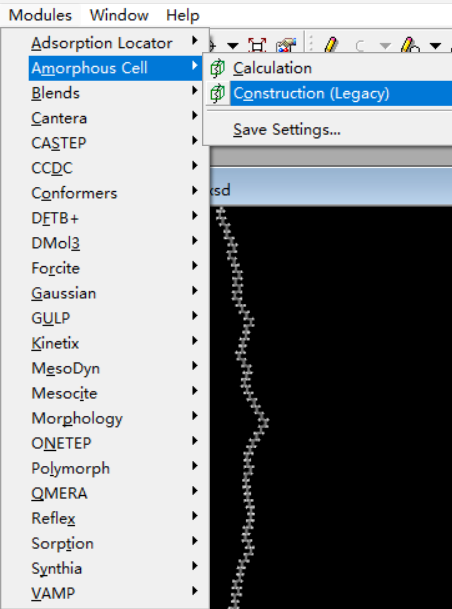
c 建盒子
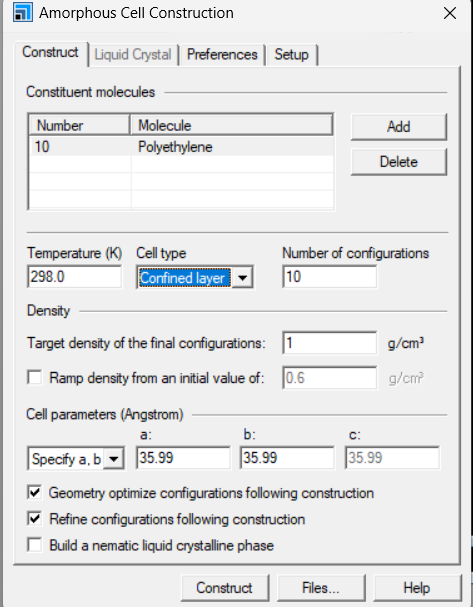
AC模块下的construction(Legacy),确保优化后的PE单链界面处于打开状态,进行add,选择cell type,设置密度,盒子大小等。这里我选用的是confined layer,至于和periodic有什么区别,自己可以进行尝试。设置numbers of configuration框为1,要不然你电脑会特别卡,然后取消下面两个勾选框。Setup栏注意力场要和之前的力场保持一致,后续的所有过程都要检查力场。
d 进行能量最小化
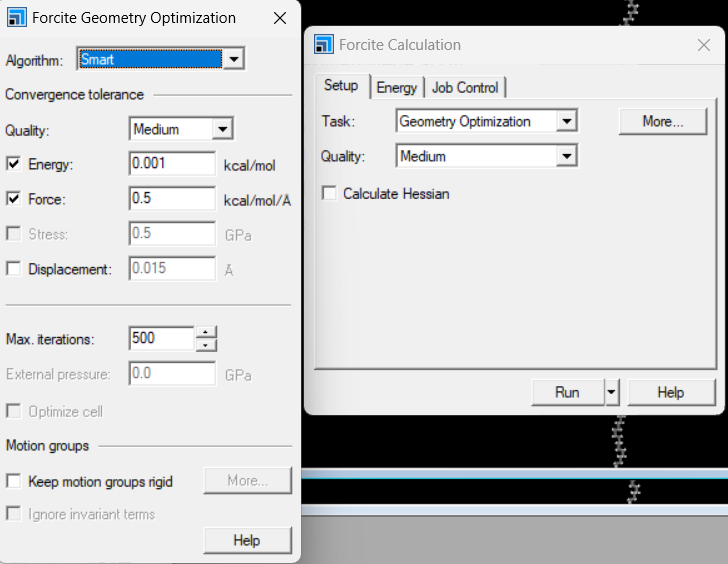
和步骤b类似,为了后续的弛豫过程不报错,在这里可以多进行几次结构优化,Steepest descent, Conjugate gradient, Quasi-Newton各来一次。
e 弛豫。
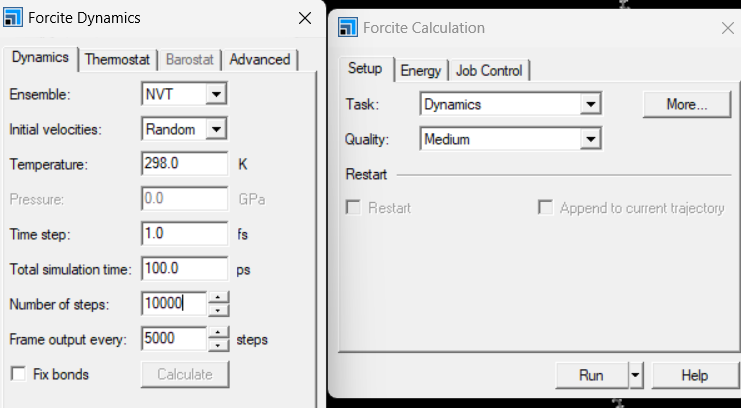
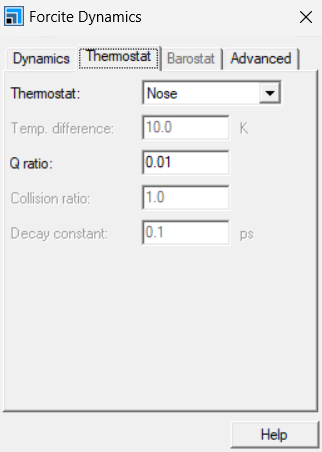
同理,将需要优化后的结构显示与主屏幕,FC中选择Dynamcis,检查力场,在more中选择系综,个人在MS中一般用NVT,弛豫时间100ps。热浴为经典的Nose-Hoover,确认力场,其余默认,弛豫时间一般较长,看电脑性能。
e 导出模型
点击打开最后的xtd文档,然后点击export导出你想要的格式就可以了,这里我们导出PDB格式
二 GROMACS的使用
参考文章
https://mp.weixin.qq.com/s/LiWO1F199Pxy3eLUFN7jhA
https://jerkwin.github.io/2017/09/14/GROMACS非标准残基教程1-修改力场与增加残基/
官方教程文档:http://www.mdtutorials.com/gmx/membrane_protein/03_solvate.html
参数内容:https://www.compchems.com/gromacs-mdp-file-parameters/#mdp-file-for-npt-equilibrations
参数官方文档:https://manual.gromacs.org/current/user-guide/mdp-options.html
中文官方教程(感觉是偏原理解释,没有代码操作教程):https://jerkwin.github.io/GMX/GMXman-1/
1 拓扑文件准备:
只有构型文件是不够的,因为构型文件里面只有这些分子的各个原子的顺序和坐标,并没有它们之间的连键情况等,故进行模拟还需要体系中各成分的拓扑文件。对于蛋白质的拓扑文件,可以在gromacs中通过命令pdb2gmx来生成,因为gromacs中包含很多种氨基酸的拓扑文件和转换脚本。对于其他分子的拓扑文件,在下载构型文件时,往往会包含相应的itp文件,即为分子的拓扑文件。水分子和离子等小分子的拓扑文件在gromacs的立场里面都有,可直接调用。
2 参数文件
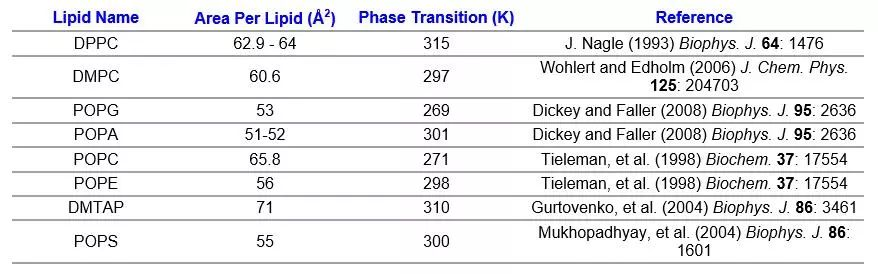
一般在gromacs tutorial中都可以直接下载(后缀为mdp的文件,包括ions.mdp, minim.mdp, nvt.mdp, npt.mdp, md.mdp),然后设置一些参数,包含模拟方法、时间、步长、输出时间间隔、键参数设置、相邻单元、静电、温度、压力、速度、周期边界条件等,有时还包括一些限制、电场、力等。对于温度的设置,应当设置在磷脂的相转变温度以上,如下是一部分磷脂的相转变温度。
也可以
3 流程
a.制作拓扑文件(化合物较小)
注意如果你是重复型的化合物坚持用tpp也可以得到拓扑但是会在后面出现问题详情在3.7有讲解
tppmktop是获得有机分子OPLSAA力场拓扑文件的好工具, 但也存在一定的不足. 一是不支持周期性分子, 二是提供的网络服务同时只能运行一个任务. 为此, 我觉得还是有必要自己做一个简单OPLSAA力场拓扑生成器GMXTOP, 一则可用于理解力场拓扑的生成方法, 二则可用于检查其他方法得到的拓扑是否合适.
参考文章:https://jerkwin.github.io/2017/09/14/GROMACS非标准残基教程1-修改力场与增加残基/
将你上面得到的聚合物pdb文件提交到这个网站上http://erg.biophys.msu.ru/erg/tpp/
然后等待一到三分钟就能得到该文件的itp和rtp文件,这两个文件是力场的拓扑文件
这个是PDB文件,这里有一个残基的名称
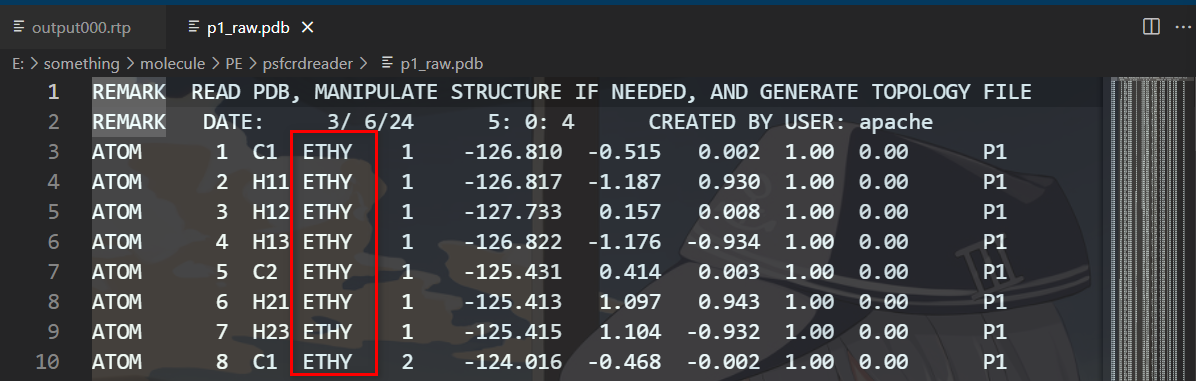
然后把导出的rtp和itp文件中的这个位置改成你残基的名称
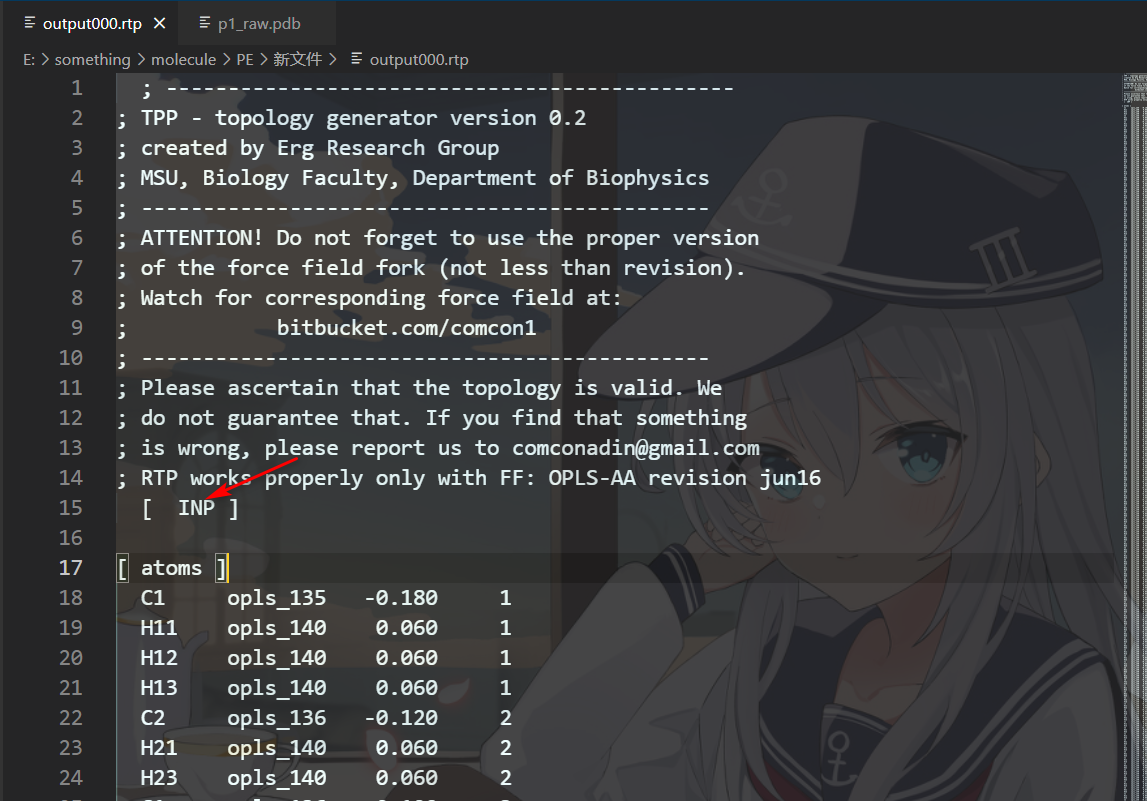
rtp和ipt文件编辑完成以后,把文件复制到**/gmx2024.1/share/gromacs/top/oplsaa.ff** 的文件夹下,然后进行下面的操作。
b.制作拓扑文件(化合物较大并重复)
参考文章:http://bbs.keinsci.com/thread-41715-1-1.html
进入网站:https://cloud.hzwtech.com/web/personal-space/auto-ff/all-atom
上传你的文件并选择你想要的立场等参数就可以生成对应的文件辣,这个网站实在是太方便了
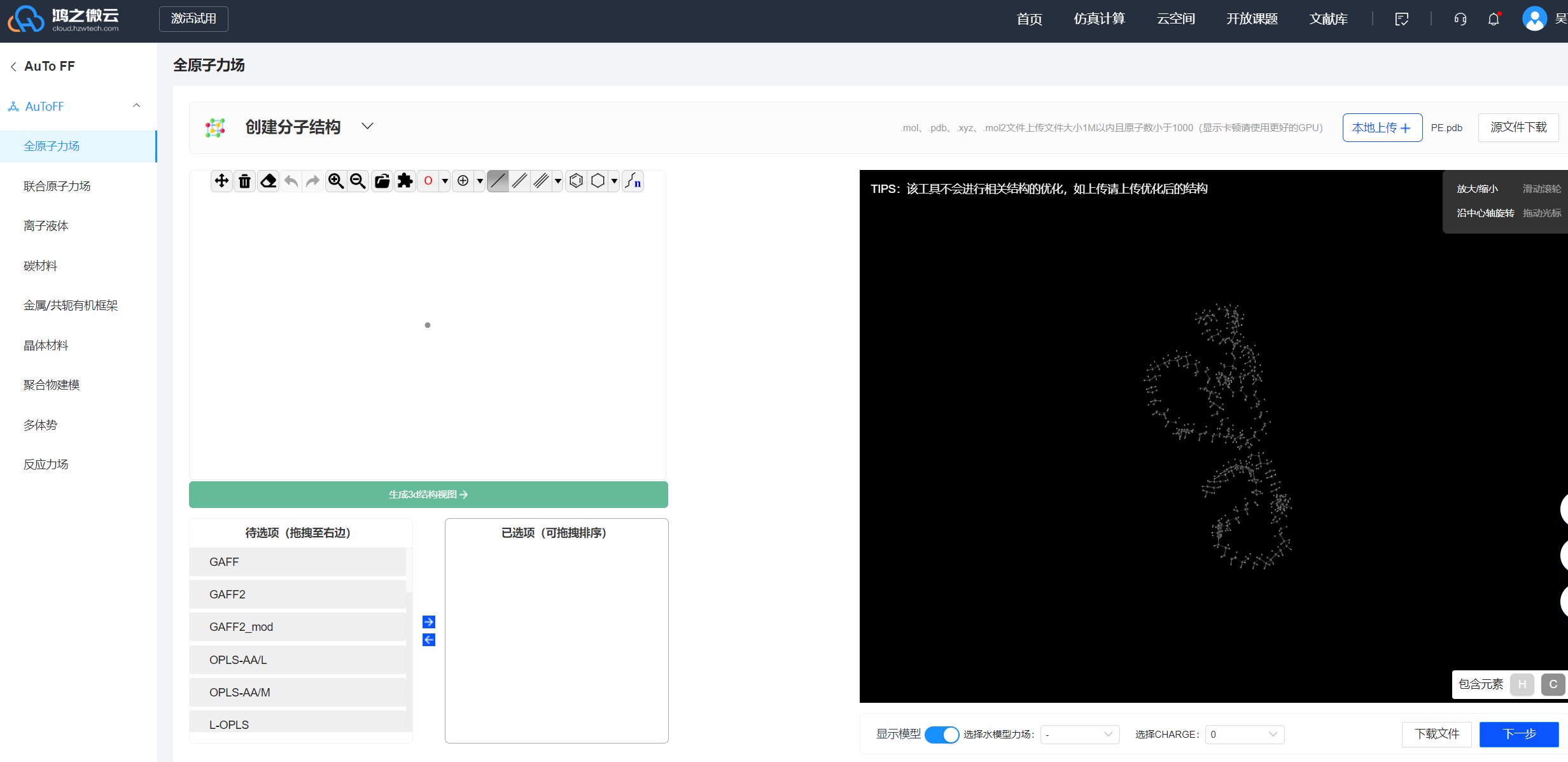
c. 构造体系
(1)将多肽的pdb文件转换成gro文件,同时生成其拓扑文件。(如果你进行了上面拓扑这一步可跳过,直接使用上面生成的gro文件)
1 | gmx pdb2gmx –f protein.pdb –o protein.gro |
在模拟蛋白质的时候,要考虑氢原子、蛋白质的两端以及带电残基等,可以用-ingh(忽略氢原子), -ter(选择多肽的两端氨基和羧基的带电情况), -inter(交替分配电荷状态)来实现。在这一步中将会选择立场,蛋白质的两端。
完成该步之后生成protein.gro, topol.top, posre.itp,其中topol.top为多肽的拓扑文件,在其中引入了立场,多肽的原子性质,连键情况、形成的角等,并且引入了水和离子的拓扑文件;posre.itp是蛋白质的重原子,用于后面对蛋白质进行位置限制。实际上总的拓扑文件里只要包含立场、各个分子的结构性质即可,无论是直接写在里面也好,还是通过include来引入也好。
(2)给生成的多肽构型定义一个盒子(根据整个体系的盒子尺寸来构造),并且将多肽放在合适的位置上。
1 | gmx editconf –f protein.gro –o pro_newbox.gro –box 长 宽 高 -center 长 宽 高 |
也可以用我的默认参数
1 | gmx editconf -f protein.gro -o pro_newbox.gro -c -d 1.0 -bt cubic |
盒子的大小要取决与膜的大小(见下面步骤)。为了将多肽放在膜上方来进行模拟,所以在此定义的位置应该与后面生物膜的位置有一定距离。该步生成pro_newbox.gro。
(3)给生物膜定义一个同样的盒子,并放置在相同的位置上。
1 | gmx editconf –f bilayer.pdb –o bilayer_newbox.gro –box 长 宽 高 -center 长 宽 高 |
注意双层膜的位置不要与多肽重叠。并且盒子的长和宽要与膜的大小相匹配,否则后面加水后磷脂的疏水尾部也将暴露在水中。如果不知道下载的膜的长宽,可以先根据经验定义一个盒子,然后用vmd打开后,输入指令pbc box,就可以显示盒子的框架了,看看是否适合,不合适就进行调整。关于vmd的使用(此处VMD教程在后面),在本文的最后会有详细介绍。如果下载的磷脂膜模型较小,可以用cat命令将两个放在一起,具体使用方法类比下面cat命令介绍。或者用如下命令:
1 | gmx genconf -f bilayer_newbox.gro -nbox 2 1 1 -o bilayer_big.gro |
其中-nbox后面的数字分别是在x、y、z方向上复制的倍数。得到的新的磷脂膜最好经过一段时间的平衡,使其连接处更加连贯。
(4)将多肽与膜放在一起,并且添加水分子。
1 | cat pro_newbox.gro bilayer_newbox.gro > whole.gro |
其中这里只是把两个分子强行融合到一个盒子里面,难免会有误差比如这样

因此我们需要通过软件来调整PE的位置,因为gro文件格式如下,我们在Excel里面就可以调整我们想要调整分子的坐标,但是这样不直观,每次的改变都不能看到,因此我们是用VMD进行调整。具体调整过程请看4.a
我们把CHARMM-GUI生成的DPPC文件中的top文件包含进我们上面的top文件里,
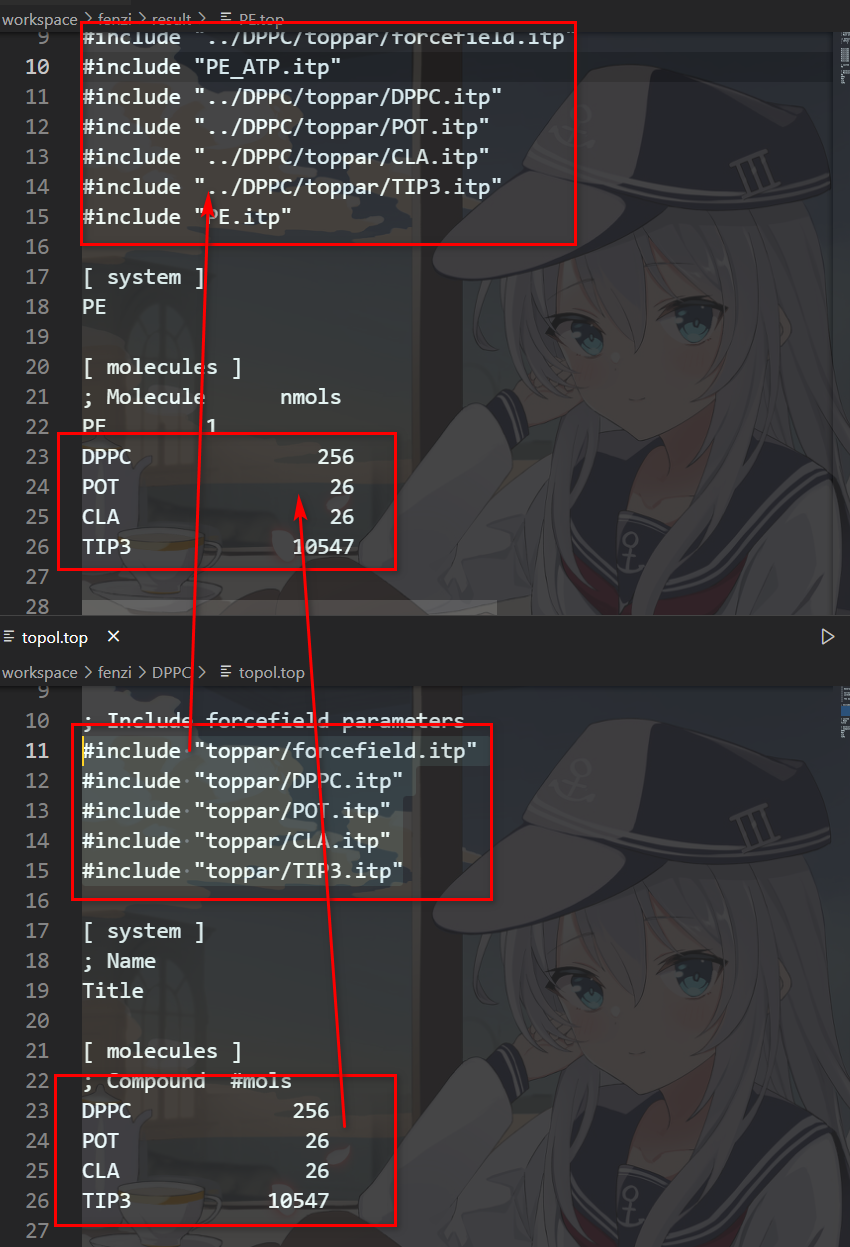
然后一定要按顺序导入文件
1 | #include "../DPPC/toppar/forcefield.itp" |
第一第二行是分子的定义,一定一定要把分子定义放在前,不然会报Invalid order for directive 的错误。
什么是分子的定义,含有[atomtypes]标签的就算作分子的定义
这段实在没懂咋设置我们实验的值在加水之前,应当将vdwradii.dat中的C的值从0.15改为0.375,这样是为了防止加水后生物膜的空隙中也有水,如果此时还有极少量的水分子在膜空隙中,可以用写字板等直接编辑生成的gro文件,删除其中的水分子,具体步骤祥见下面的vmd部分。删除水分子之后,别忘了top文件里面相应的水分子数目要减少。
注意:如果之前DPPC加过水了,这里就不用加水
1 | gmx solvate –cp whole.gro –cs spc216.gro –o sol.gro –p topol.top |
d 开始模拟
(1)添加约束力(可跳过)(后面也有约束力)
1 | gmx genrestr -f sol.gro -o strong_posre.itp -fc 100000 100000 100000 |
选择整个系统0
在用于最小化的 mdp 文件,添加一行“ Definition =-DSTRONG _ POSRES”来使用这些新的位置约束。
inflategro.pl下载路径如下:http://www.mdtutorials.com/gmx/membrane_protein/Files/inflategro.pl
原文语句:perl inflategro.pl system.gro 4 DPPC 14 system_inflated.gro 5 area.dat
- 4-应用的缩放因子。值 > 1表示通货膨胀,值 < 1表示收缩/压缩
- DPPC-用于脱皮的油脂的残留名称
- 用于搜索要删除的脂质的截止半径(单位: Å)
- 每个油脂计算面积的5个网格间距(也以 Å 为单位)
- Dat-输出文件,包含每个脂质的面积信息,可用于评估结构的适应性
因此语句为
1 | perl inflategro.pl sol.gro 1 POPE 12 system_inflated.gro 5 area.dat |
然后会被删除一些原子因此我们需要再topol.top中的molecules 模块更改修改后的原子数目。
(2)能量最小化文件准备
在每个不同的体系下需要不同的参数,这里面以蛋白质为例,你可以根据你想要的参数进行调整
1 | ; LINES STARTING WITH ';' ARE COMMENTS |
1 | gmx grompp –f minim.mdp –c system_inflated.gro –p topol.top -r system_inflated.gro –o min.tpr |
如果这里又报错可以强行跳过
-maxwarn 6
输出文件包括构型文件(min.gro)、日志文件(min.log)、轨迹文件(min.trr、min.xtc)。可以通过修改参数文件内的输出时间间隔来控制输出,最好不要过于细致(占内存较大),也不要过于粗略(不利于分析)。
如果这一步报错:No default Proper Dih. types 移步到3.3
然后运行
1 | gmx mdrun -v -deffnm min |
如果系统没有收敛则移步:http://www.mdtutorials.com/gmx/membrane_protein/05_EM.html
如果报错:The [molecules] section of your topology specifies more than one block of a [moleculetype] with a [settles] block. 移步3.6
输出文件包括构型文件(min.gro)、日志文件(min.log)、轨迹文件(min.trr、min.xtc)。可以通过修改参数文件内的输出时间间隔来控制输出,最好不要过于细致(占内存较大),也不要过于粗略(不利于分析)。
(3)建立index文件/约束力
1 | gmx make_ndx -f min.gro -o index.ndx |
在建立过程中,会选择分组,比如,我们可以将水和离子分为一组,根据提示可以将水和离子前面的序号用“|”连接即可,如14|16。
1 | PE_DPPC |
然后用q退出
热平衡和压力平衡,为了防止在NVT和NPT的时候蛋白复合物爆开,所以要加一个力·的限制,首先构建配体的力限制
1 | #创建索引文件 |
给增加1000的三个方向的力
1 | gmx genrestr -f min.gro -n index_force.ndx -o force.itp -fc 1000 1000 1000 |
之间插入位置
1 | ; Include ligand Position restraint file |
(4)nvt模拟
nvt.mdp
1 | define = -DPOSRES -DPOSRES_FC_LIPID=1000.0 -DDIHRES -DDIHRES_FC=1000.0 ; position restrain the protein and ligand |
注意tc-grps = PE_DPPC POT_CLA_TIP3的热浴组合温度需要改成自己的,这个体系用的常温300K,跑的时间为100ps
1 | gmx grompp -f nvt.mdp -c min.gro -r min.gro -p topol.top -n index.ndx -o nvt.tpr |
nvt平衡中通常使用的热浴为Berendsen或者V-rescale,使最终温度能基本达到稳定。
类似能量最小化,只须将脚本中的min改为nvt即可。输出文件比能量最小化多了一个断点文件(nvt.cpt)
完事以后我们继续对产生的文件进行
1 | gmx mdrun -v -deffnm nvt |
(5)npt模拟
nvt模拟之后温度基本达到稳定,但是还需将模拟盒子根据设定的压力进行平衡模拟以松弛磷脂膜,因此进行npt模拟只需限制蛋白质即可。但还应注意的是,在npt模拟中一般使用Nose-Hoover热浴,与nvt中的两个热浴不同,该热浴允许比较大的涨落,因此不适用于nvt模拟。对于压力运用Parrinello-Rahman方法,由于模拟盒子中有磷脂膜,所以我们在此使用semiisotropic,与膜平行的两个方向为一个压力,与膜垂直的方向为一个压力。
npt.mdp
1 | title = Protein-ligand complex NPT equilibration |
1 | gmx grompp –f npt.mdp –c nvt.gro -r nvt.gro –t nvt.cpt –p topol.top –n index.ndx –o npt.tpr |
将脚本中nvt改为npt,即可提交任务。
1 | gmx mdrun -v -deffnm npt |
(6)md模拟
平衡了温度和压力之后,开始较长时间的md模拟,模拟过程与npt类似。
如果计算结束以后,觉得仍未平衡,或者想要更长时间的模拟,可以进行续算,一种方法是类似上面,只是将md.gro作为初始构型,进行md模拟;另一种方法是延长计算时间,用-extend 10000延长10000ns。
md.mdp
1 | title = Protein-ligand complex MD simulation |
1 | gmx grompp -f md.mdp -c npt.gro -r npt.gro -t npt.cpt -p topol.top -n index.ndx -o md_0_1.tpr |
1 | gmx mdrun -deffnm md_0_1 #不使用GPU加速或者无GPU运行 |
(100ns使用1660sGPU加速跑了30+个小时,如果用CPU的话可能要500+小时,所以建议使用GPU加速)
e 分析(可选)
关于分析,gromacs具有很多命令,可以直接进行分析,可根据模拟需求来进行选择。在分析之前,应当先建立分组索引文件,再对其中的某一组或者几组进行分析,包括能量(gmx energy)、密度(gmx density)、径向分布函数(gmx rdf)、均方位移(gmx msd)、氢键(gmx hbond)、回旋半径(gmx gyrate)、蛋白质的二级结构(gmx do_dssp)、静电势(gmx potential)等。同时,gromacs还提供了查看轨迹文件工具 gmx view,此方法无需OpenGL支持。比如对于磷脂膜密度的分析,在做分析之前要建立相应的index文件:
1 | gmx make_ndx -f md.tpr -o density_groups.ndx |
选择的原子取决于磷脂的构型。
更多的分析可参见http://www.mdtutorials.com/gmx/membrane_protein/09_analysis.html
f 显示
着色方法调试:http://course.sdu.edu.cn/Download2/20151113131122124.pdf
我们把跑出来的结果进行可视化展示,我们把轨迹文件转换成PDB文件放到VMD里面进行展示,可以看出来我们跑出来的trr文件特别大,相比较来说xtc文件较小,因此我们最后可以只保留xtc文件,并对结果进行抽帧,我是模拟100ns,所以每隔1ns抽一帧,组成新的轨迹文件,放到PDB里面打开。
1 | ❯ ls -l --block-size=g |
1 | gmx tejconv -s md_0_1.tpr -f md_0_1.xtc -o traj.pdb -dt 1000 |
如果使用VMD观看时有胡乱的线连接,则使用如下方法
参考:http://bbs.keinsci.com/thread-20047-1-1.html
- 用gmx trjconv命令接-pbc mol转换轨迹,使分子保持完整
- 用Dynamic Bonds绘制风格显示
- 命令行窗口执行mol bondsrecalc all;topo retypebonds;(只解决当前一帧的问题)
4 VMD下载使用(win)
在这里下载并安装,需要注册,随便注册一个账户就行。
https://www.ks.uiuc.edu/Development/Download/download.cgi?PackageName=VMD
安装完一共有以下三个界面
我们先把想要显示的文件(PDB)拖拽到主窗口处,注意路径一定要是英文的,不然会有乱码
一般来说会自动在窗口处显示,如果显示过小想调整器大小和连接,网上有各种教程。
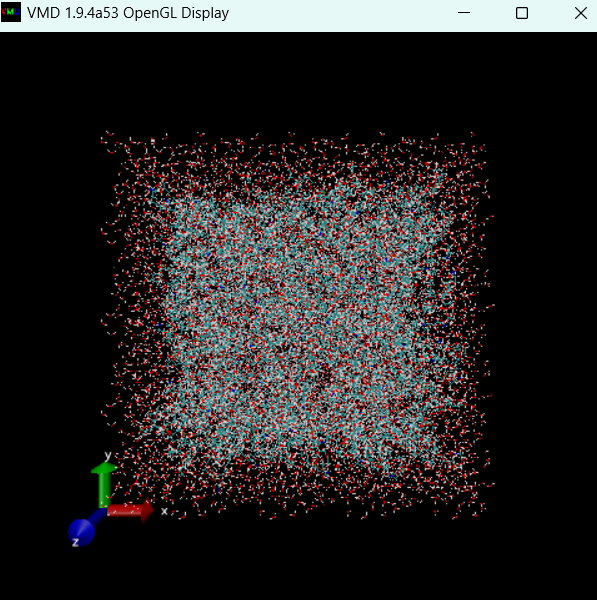
我们使用以下命令就可以确定分子的边界
1 | pbc set {长 宽 高 } |
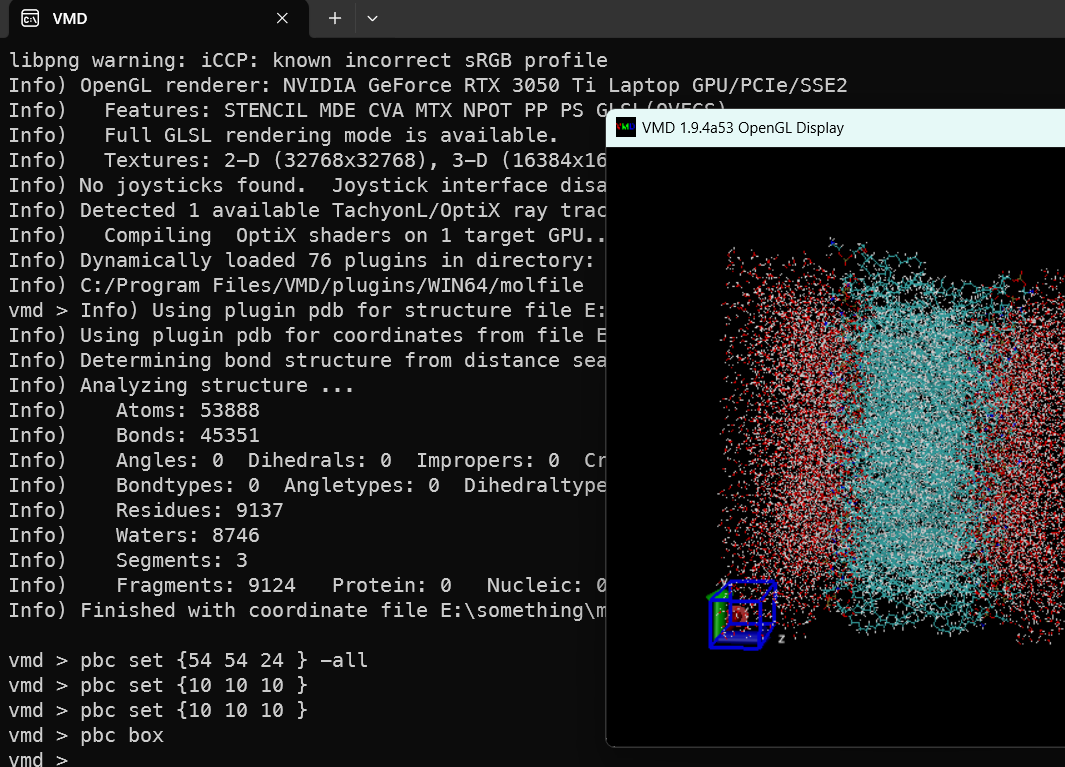
a 调整部分分子的位置
我们先更改我们的背景颜色
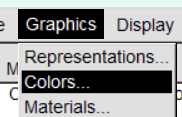
我们改成白色
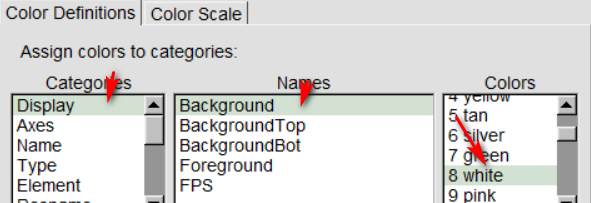
我们先调整整体视图的样式,这样可以使我们更清晰的看到我们想调整的分子。
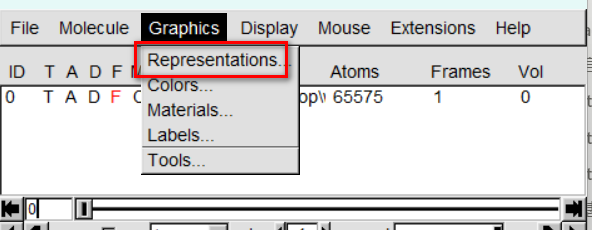
在我构成的gro文件中,一共主要分为以下三个部分:water,DPPC,PE
因此我想要分层展示,点击create rep。
Singlewords指的是对一整个化学化合物的分类,比如蛋白质,聚合物,水等等,这些不因你导入的文件改变而改变
下面Keyword一栏指的是对你导入的pdb或者gro文件中分子的分类,比如resname就是对你文件中残基进行分类的。
如果你想选取你想要的分子,按照如下格式填写即可,其中Singlewords直接输入即可。
并且这里分子的选取还提供集合操作,并and交or非not,按照你想选取的分子进行输入即可
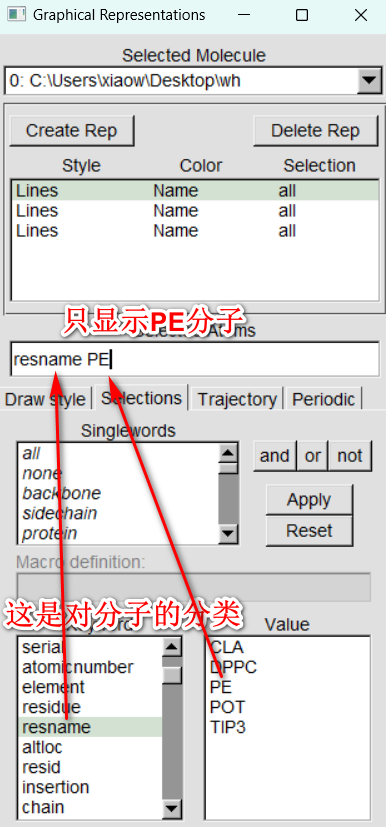
接下来我们可以对每一个分类后的分子进行不同程度的展示染色,这是我调整后的视图,双击你分类的分子即可隐藏
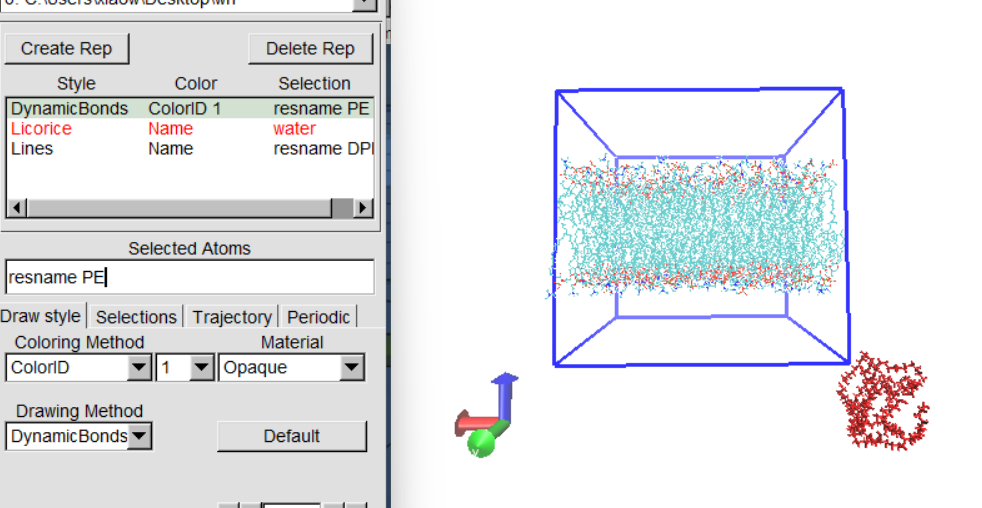
接下来我们对分子进行移动
参考:https://www.ks.uiuc.edu/Research/vmd/vmd-1.7.1/ug/node108.html
https://www.ks.uiuc.edu/Research/vmd/vmd-1.7.1/ug/node181.html
在VMD命令行中操作
因为我们要移动PE分子,因此我们需要先选中所有PE中的原子,然后对每一个分子的坐标进行加减操作。
1 | set sel [atomselect top "resname PE"] #导入我们选中的原子 sel是我们储存的变量 |
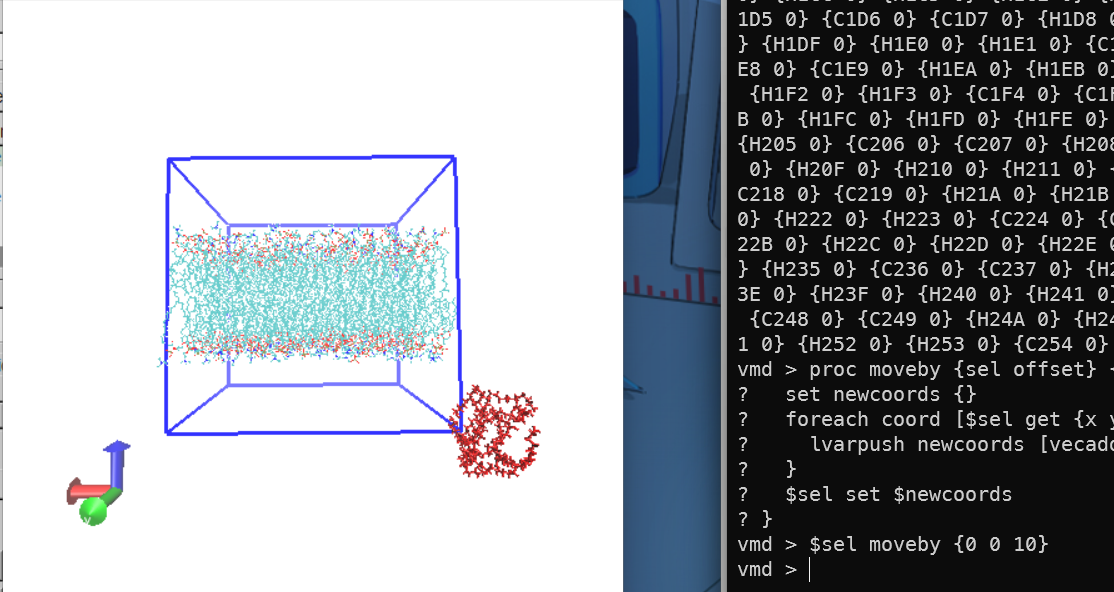
以此类推你就可以得到你想要的位置,达到你想要的位置以后就可以右键点击保存为你想要的格式了
三 、绕过的弯路和知识
1 报错:Residue ‘XXX’ not found in residue topology databases
报错找不到残基,在工具的官网上终于找到解释了
连接如下https://manual.gromacs.org/current/reference-manual/functions/force-field.html
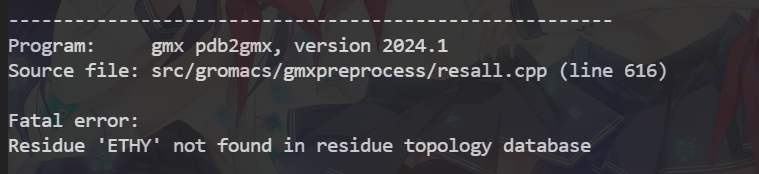
PP/PE/PET属于聚合物,不适用于提供的力场,就是在力场文件中找不到对应的残基在2.3.a小节中制作拓扑文件即可完成。因为
%20d0e4e4faba0447f6895cb79f67055395/Untitled%2035.png)
然后参考这篇文章https://zhuanlan.zhihu.com/p/78912262
2 报错No default Proper Dih. types
解决帖子:https://gromacs.bioexcel.eu/t/dih-types-error-in-ions-adding-steps-in-gromacs/3914
原因是当时引入分子的拓扑文件时没有引入itp文件,只引入rtp文件导致读取依赖的Dih找不到所导致的。因此把上面的两个拓扑文件引入则问题解决
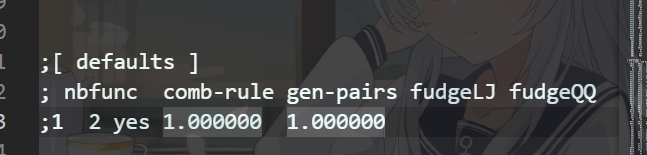
3 报错Syntax error - File forcefield.itp, line 13
这个是因为 forcefield.itp里面的defaults与其他文件里面的defaults冲突导致的报错,因此我们注释掉这个语句即可
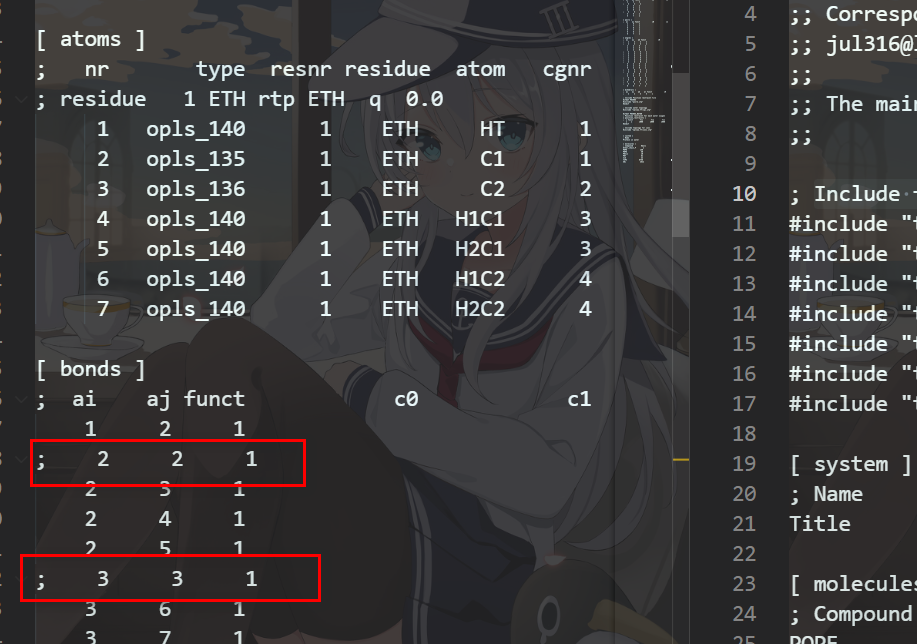
4 报错Duplicate atom index (3) in angles
但凡出现Duplicate atom index (X) in XXX 原因就是数字重复。比如如下例子,两个原子链接,这里是不允许两个相同的原子相连的。因此把报错的部分注释掉即可
5 报错Cannot find position restraint file restraint.gro (option -r).
在GROMACS 2018版本以后,为了避免错误,需要明确地指定位置约束坐标文件,教程比较老了,基本是17,18年的教程,因此原教程没有这一步。2.3.b.1加了这一步。
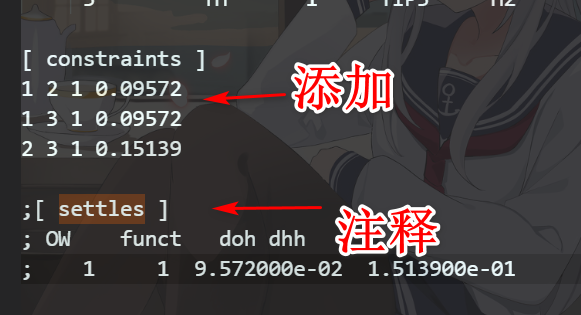
6 The [molecules] section of your topology specifies more than one block of a [moleculetype] with a [settles] block.
解决博客:https://gromacs.bioexcel.eu/t/solvent-partition/1034
这是因为settles的定义报错,因此把settles注释掉换成角度就可以了,我在网上看到基本所有的settles的报错都是来自TIP3.itp 中的这里
1 | [ constraints ] |
7 重复型聚合物运行TPP
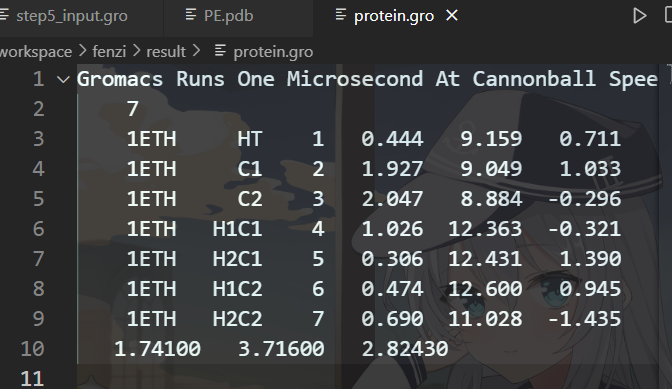
这里使用的是PE100(C202H404)。PE.pdb,应该有六百多个分子的但是经过2.3.c.1后会得到如下图结果
1 | gmx pdb2gmx –f protein.pdb –o protein.gro |
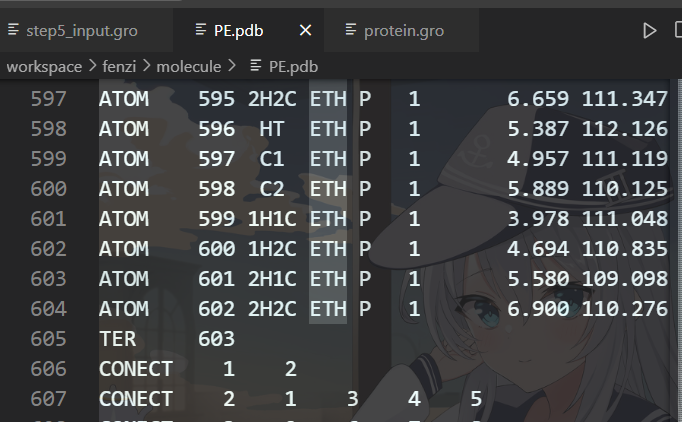
只会把头基的7个分子记录在里面,并且在后续的计算中我发现真的就只有这7个分子,也不知道哪里出了问题,反正是会导致这个问题,有没有大佬可以解答一下
8 拓扑文件有问题
因为分子定义的顺序问题导致的错误,这个网站是可以使用的!!!
这个网站下载后的文件我没太搞懂咋弄的,后面一直在报错top有问题,好像是因为立场不同的原因。
参考文章:https://jerkwin.github.io/2017/06/29/GMXTOP-OPLSAA力场的GROMACS拓扑文件生成器/
感谢作者的贡献,可以直接成成top和gro文件
登录以下界面,加载你的PDB文件,然后点击AUTO,然后点击create,然后就可以下载你想要的文件辣
https://jerkwin.github.io/prog/gmxtop.html



